方案详情
文
Thermal shift assay(TSA),也被称为差示扫描荧光法(DSF)是表征蛋白热稳定性的常用方法之一,广泛应用于蛋白配体互作表征,突变体、缓冲液、去垢剂筛选等领域。但DSF的实验操作较繁琐,需要根据蛋白的特性及去垢剂兼容性选择合适的染料,优化蛋白和染料的比例,在配制样品时还要考虑染料自带的有机溶剂对蛋白的影响。
时下被行业深度认可的无标记的TSA验证方法-也称nanoDSF技术,可解决DSF技术的局限性,样品无需加染料就可以直接上机检测了。下面我们一起通过用户的文献案例来进一步了解。
方案详情

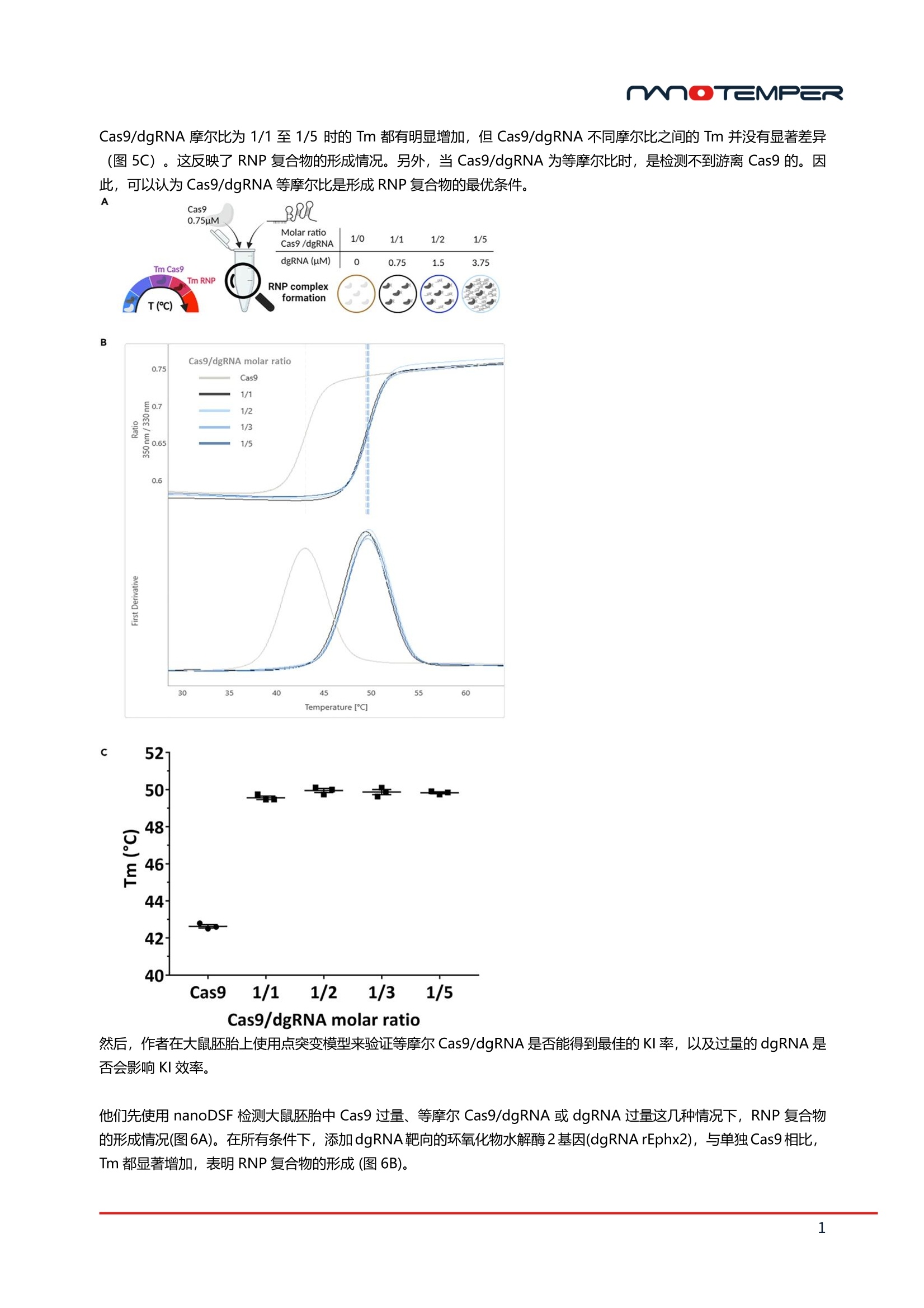
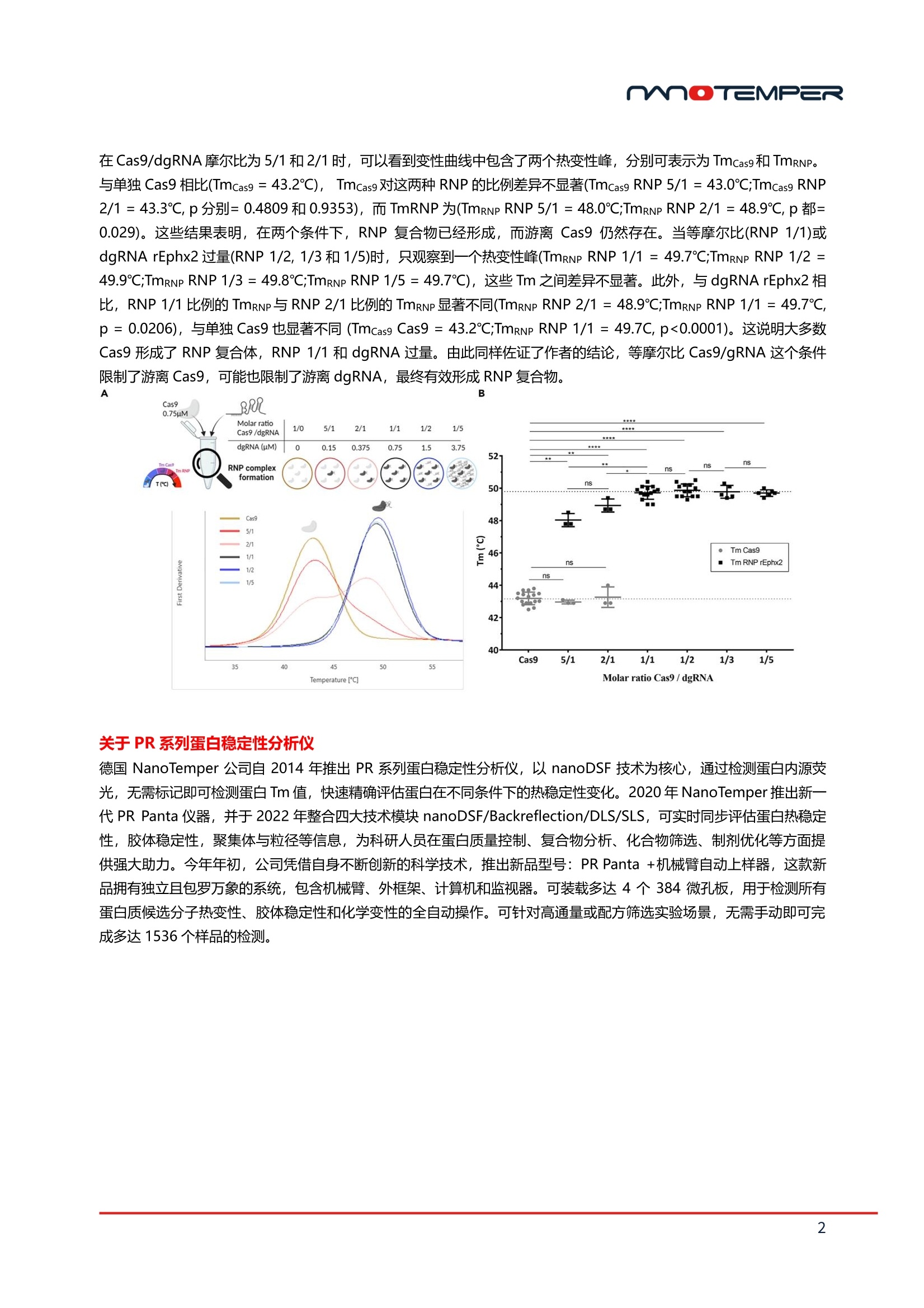
01前言Thermal shift assay(TSA),也被称为差示扫描荧光法(DSF)是表征蛋白热稳定性的常用方法之一,广泛应用于蛋白配体互作表征,突变体、缓冲液、去垢剂筛选等领域。但DSF的实验操作较繁琐,需要根据蛋白的特性及去垢剂兼容性选择合适的染料,优化蛋白和染料的比例,在配制样品时还要考虑染料自带的有机溶剂对蛋白的影响。替代性技术:nanoDSF技术时下被行业深度认可的无标记的TSA验证方法-也称nanoDSF技术,可解决DSF技术的局限性,样品无需加染料就可以直接上机检测了。下面我们一起通过用户的文献案例来进一步了解。02NanoTemper用户应用案例解读检测样品:Cas9,gRNA使用仪器:PR系列蛋白稳定性分析仪涉及技术:nanoDSF技术https://doi.org/10.1016/j.isci.2023.106399CRISPER - Cas9介导的基因编辑能够帮助人们在动物和细胞模型中实现广泛的靶向敲除(KO)或敲入(KI),例如单点或多点KO、点突变、报告基因KO或KI等。然而,CRISPR-Cas9的切割效果和准确性仍是基因编辑面临的主要挑战,它们特别受到核糖核蛋白复合物 (RNP)的组装配比的影响。2023年3月,法国南特大学的研究人员近期发表了研究成果:Excess of guide RNA reduces knockin efficiency and drastically increases on-target large deletions,作者借助PR系列蛋白稳定性分析仪搭载的nanoDSF技术,一种无需标记和固定化的方法,证明了Cas9和gRNA的等摩尔比是形成RNP复合物的最佳条件。研究结果作者通过CRISPR/Cas9 KI将双等位纯合子GFP hiPSCs转化为BFP表达细胞,通过细胞表型分析证明在Cas9的最佳浓度为0.4 mM时,增加ssODN浓度有助于降低KO率,提高KI率,而增加dgRNA对KO和KI没有影响。0.4 mM的Cas9、等摩尔Cas9/gRNA比例和2 mM的ssODN是在hiPSC中实现高效GFP到BFP转换的最佳选择。 在此之前,有一些研究曾表明过量的gRNA对于靶向切割有帮助,而本文作者的实验结果则与之产生了矛盾。于是作者使用NanoTemper公司的nanoDSF技术建立了一套体外实验方法学,用来评估RNP复合物的形成效率,从而佐证他们的结论。通过nanoDSF这种无标记技术,可在升温过程中检测蛋白中的色氨酸和酪氨酸的自发荧光。随着升温,仪器同步采集在350 nm和330 nm处的最大发射光位移,并自动拟合出蛋白的热展开曲线。NanoDSF能够检测蛋白的构象变化导致的热稳定性差异,并由熔解温度(Tm)来表征蛋白的热稳定性。由于Cas9是一种含有色氨酸和酪氨酸残基的变构酶, 因此利用nanoDSF技术可以简单、直观地检测导致Cas9重排的RNP复合物的形成。 为了确定hiPSC中使用的dgRNA靶向GFP位点(dgRNA GFP)形成RNP复合物的有效性,作者使用恒定浓度的Cas9 (0.75 mM)与一定浓度范围内的dgRNA, 通过nanoDSF检测,作出升温诱导的蛋白变性曲线检测 (图1A)。根据一阶导数,可以确定Cas9单独的Tm(TmCas9 = 43.2C),以及Cas9/dgRNA摩尔比为1/1至1/5 的Tm:1/1 时TmRNP = 49.6 C、1/2 时TmRNP = 50.0 C、1/3时 TmRNP = 49.9C、1/5 时TmRNP = 49.8 C (图1B)。相比只有Cas9时, Cas9/dgRNA摩尔比为1/1至1/5 时的Tm都有明显增加,但Cas9/dgRNA不同摩尔比之间的Tm并没有显著差异(图1C)。这反映了RNP复合物的形成情况。另外,当Cas9/dgRNA为等摩尔比时,是检测不到游离Cas9的。因此,可以认为Cas9/dgRNA等摩尔比是形成RNP复合物的最优条件。图1作者在大鼠胚胎上使用点突变模型来验证等摩尔Cas9/dgRNA是否能得到最佳的KI率,以及过量的dgRNA是否会影响KI效率。 他们先使用nanoDSF检测大鼠胚胎中Cas9过量、等摩尔Cas9/dgRNA或dgRNA过量这几种情况下,RNP复合物的形成情况(图2A)。在所有条件下,添加dgRNA靶向的环氧化物水解酶2基因(dgRNA rEphx2),与单独Cas9相比,Tm都显著增加,表明RNP复合物的形成 (图2B)。 在Cas9/dgRNA摩尔比为 5/1和2/1时,可以看到变性曲线中包含了两个热变性峰,分别可表示为TmCas9和TmRNP。与单独Cas9相比(TmCas9 = 43.2℃), TmCas9对这两种RNP的比例差异不显著(TmCas9 RNP 5/1 = 43.0℃;TmCas9 RNP 2/1 = 43.3℃, p分别= 0.4809和0.9353),而TmRNP为(TmRNP RNP 5/1 = 48.0℃;TmRNP RNP 2/1 = 48.9℃, p 都= 0.029)。这些结果表明,在两个条件下,RNP复合物已经形成,而游离Cas9仍然存在。当等摩尔比(RNP 1/1)或dgRNA rEphx2过量(RNP 1/2, 1/3和1/5)时,只观察到一个热变性峰(TmRNP RNP 1/1 = 49.7℃;TmRNP RNP 1/2 = 49.9℃;TmRNP RNP 1/3 = 49.8℃;TmRNP RNP 1/5 = 49.7℃),这些Tm之间差异不显著。此外,与dgRNA rEphx2相比,RNP 1/1比例的TmRNP与RNP 2/1比例的TmRNP显著不同(TmRNP RNP 2/1 = 48.9℃;TmRNP RNP 1/1 = 49.7℃, p = 0.0206),与单独Cas9也显著不同 (TmCas9 Cas9 = 43.2℃;TmRNP RNP 1/1 = 49.7C, p<0.0001)。这说明大多数Cas9形成了RNP复合体,RNP 1/1和dgRNA过量。由此同样佐证了作者的结论,等摩尔比Cas9/gRNA这个条件限制了游离Cas9,可能也限制了游离dgRNA,最终有效形成RNP复合物。图203关于PR系列蛋白稳定性分析仪德国NanoTemper公司自2014年推出PR系列蛋白稳定性分析仪,以nanoDSF技术为核心,通过检测蛋白内源荧光,无需标记即可检测蛋白Tm值,快速精确评估蛋白在不同条件下的热稳定性变化。2020年NanoTemper推出新一代PR Panta仪器,并于2022年整合四大技术模块nanoDSF/Backreflection/DLS/SLS,可实时同步评估蛋白热稳定性,胶体稳定性,聚集体与粒径等信息,为科研人员在蛋白质量控制、复合物分析、化合物筛选、制剂优化等方面提供强大助力。今年年初,公司凭借自身不断创新的科学技术,推出新品型号:PR Panta + 机械臂自动上样器,这款新品拥有独立且包罗万象的系统,包含机械臂、外框架、计算机和监视器。可装载多达4个384微孔板,用于检测所有蛋白质候选分子热变性、胶体稳定性和化学变性的全自动操作。可针对高通量或配方筛选实验场景,无需手动即可完成多达1536个样品的检测。PR系列产品mOTEMPER M O TEMPER 使用无标记 TSA 方法评估 CRISPER-Cas9 RNP 复合物形成的最佳条件 用户为什么选择无标记的 TSA 验证方法? 它可以解决哪些问题? Thermal sh i f t assay (TSA) , 也被称为差示扫描荧光法 (DSF) 是表征蛋白热稳定性的常用方法之一,广泛应用于蛋 白配体互作表征,突变体、缓冲液、去垢剂筛选等领域。但 DSF 的实验操作较 繁 琐,需要根据蛋白的特性及去垢剂兼 容性选择合适的染料,优化蛋白和染料的比例,在配制样品时还要考虑染料自带的有机溶剂对蛋白的影响 。 而时下被行业深度认可的无标记的 TSA 验证方法-也称 nanoDSF 技术, 可解决 DSF 技术的局限性,样品无需加染料 就可以直接上机检测了。下面我们一起通过用户的文献案例来进一步了解。 NanoTemper 用户应用案例解读 原文链接: https://doi.org/10.1016/j.isci.2023.106399 检测样品: Cas9, gRNA 使用仪器:PR系列蛋白稳定性分析仪 涉及技术: nanoDSF技术 CRISPER - Cas9 介导的基因编辑能够帮助人们在动物和细胞模型中实现广泛的靶向敲除(KO)或敲入(KI),例如单点或 多点KO、点突变、报告基因 KO或KI等。然而, CRISPR-Cas9 的切割效果和准确性仍是基因编辑面临的主要挑战,它们特别受到核糖核蛋白复合物 (RNP)的组装配比的影响。 2023年3月,法国南特大学的研究人员近期发表了研究成果: Excess of guide RNA reduces knockin efficiency and drastically increases on-target large deletions, 作者借助PR系列蛋白稳定性分析仪搭载的 nanoDSF技术,一种无需标记和固定化的方法,证明了 Cas9 和 gRNA的等摩尔比是形成 RNP 复合物的最佳条件。 研究结果 作者通过 CRISPR/Cas9 KI将双等位纯合子 GFP hiPSCs 转化为 BFP表达细胞,通过细胞表型分析证明在 Cas9的最佳 浓度为 0.4mM时,增加 ssODN 浓度有助于降低KO率,提高KI率,而增加 dgRNA 对KO和KI没有影响。0.4mM 的 Cas9、等摩尔 Cas9/gRNA 比例和2 mM 的 ssODN 是在 hiPSC 中实现高效 GFP到BFP转换的最佳选择。 在此之前,有一些研究曾表明过量的 gRNA 对于靶向切割有帮助,而本文作者的实验结果则与之产生了矛盾。于是作 者使用 NanoTemper 公司的 NanoDSF 技术建立了一套体外实验方法学,用来评估RNP复合物的形成效率,从而佐证 他们的结论。 通过 nanoDSF 这种无标记技术,可在升温过程中检测蛋白中的色氨酸和酪氨酸的自发荧光。随着升温,仪器同步采集 在 350 nm 和 330 nm 处的最大发射光位移,并自动拟合出蛋白的热展开曲线。NanoDSF 能够检测蛋白的构象变化 导致的热稳定性差异,并由熔解温度(Tm)来表征蛋白的热稳定性。由于 Cas9 是一种含有色氨酸和酪氨酸残基的变构 酶,因此利用 NanoDSF技术可以简单、直观地检测导致 Cas9 重排的 RNP 复合物的形成。 为了确定 hiPSC 中使用的 dgRNA 靶向 GFP 位点(dgRNA GFP)形成 RNP复合物的有效性,作者使用恒定浓度的 Cas9(0.75mM)与一定浓度范围内的 dgRNA, 通过 nanoDSF检测,作出升温诱导的蛋白变性曲线检测(图5A)。根据一阶 导数, 可以确定 Cas9 单独的 Tm(Tmcas9 = 43.2C), 以及 Cas9/dgRNA摩尔比为1/1至1/5的Tm: 1/1时TmRNP =49.6 C、1/2 时 TmRNP = 50.0 C、1/3 时 TmRNP = 49.9C、1/5时 TmRNP= 49.8 C (图5B)。相比只有 Cas9 时, Cas9/dgRNA 摩尔比为1/1至1/5时的 Tm 都有明显增加,但 Cas9/dgRNA 不同摩尔比之间的 Tm 并没有显著差异 (图5C)。这反映了 RNP 复合物的形成情况。另外,当 Cas9/dgRNA 为等摩尔比时,是检测不到游离 Cas9 的。因 此,可以认为 Cas9/dgRNA 等摩尔比是形成 RNP复合物的最优条件。 B C 52- Cas9/dgRNA molar ratio 然后,作者在大鼠胚胎上使用点突变模型来验证等摩尔 Cas9/dgRNA 是否能得到最佳的 KI率,以及过量的 dgRNA 是 否会影响KI效率。 他们先使用 nanoDSF检测大鼠胚胎中 Cas9 过量、等摩尔 Cas9/dgRNA 或 dgRNA过量这几种情况下, RNP 复合物 的形成情况(图6A)。在所有条件下,添加dgRNA靶向的环氧化物水解酶2基因(dgRNArEphx2), 与单独Cas9相比,Tm 都显著增加,表明 RNP 复合物的形成(图6B)。 在 Cas9/dgRNA 摩尔比为 5/1和2/1时,可以看到变性曲线中包含了两个热变性峰,分别可表示为 Tmcas9和 TmRNP。与单独 Cas9 相比(Tmcas9=43.2℃), Tmcas9对这两种 RNP的比例差异不显著(TmCas9 RNP 5/1=43.0℃;Tmcas9 RNP 2/1=43.3℃, p分别= 0.4809和0.9353), 而 TmRNP 为(TmRNP RNP 5/1 = 48.0℃;TmRNP RNP 2/1=48.9℃, p都=0.029)。这些结果表明,在两个条件下, RNP 复合物已经形成,而游离 Cas9 仍然存在。当等摩尔比(RNP 1/1)或 dgRNA rEphx2 过量(RNP 1/2, 1/3和1/5)时,只观察到一个热变性峰(TmRNP RNP 1/1= 49.7℃;TmRNP RNP 1/2=49.9℃;TmRNP RNP 1/3= 49.8℃;TmRNP RNP 1/5=49.7℃), 这些Tm 之间差异不显著。此外,与 dgRNA rEphx2 相 比, RNP 1/1比例的 TmRNP与 RNP 2/1 比例的 TmRNP 显著不同(TmRNP RNP 2/1= 48.9℃;TmRNP RNP 1/1= 49.7℃,p =0.0206), 与单独 Cas9 也显著不同 (Tmcas9 Cas9= 43.2℃;TmRNP RNP 1/1 = 49.7C, p<0.0001)。这说明大多数 Cas9 形成了 RNP 复合体, RNP 1/1和 dgRNA 过量。由此同样佐证了作者的结论,等摩尔比 Cas9/gRNA 这个条件 限制了游离 Cas9, 可能也限制了游离 dgRNA, 最终有效形成 RNP复合物。 A 关于 PR 系列蛋白稳定性分析仪 德国 NanoTemper 公司自2014年推出 PR系列蛋白稳定性分析仪,以 nanoDSF 技术为核心,通过检测蛋白内源荧 光,无需标记即可检测蛋白 Tm值,快速精确评估蛋白在不同条件下的热稳定性变化。2020年NanoTemper 推出新一 代 PR Panta 仪器,并于2022年整合四大技术模块 nanoDSF/Backreflection/DLS/SLS,可实时同步评估蛋白热稳定 性,胶体稳定性,聚集体与粒径等信息,为科研人员在蛋白质量控制、复合物分析、化合物筛选、制剂优化等方面提 供强大助力。今年年初,公司凭借自身不断创新的科学技术,推出新品型号: PR Panta +机械臂自动上样器,这款新 品拥有独立且包罗万象的系统,包含机械臂、外框架、计算机和监视器。可装载多达4个384微孔板,用于检测所有 蛋白质候选分子热变性、胶体稳定性和化学变性的全自动操作。可针对高通量或配方筛选实验场景,无需手动即可完 成多达1536个样品的检测。 PR系列产品
确定


还剩2页未读,是否继续阅读?
诺坦普科技(北京)有限公司为您提供《使用无标记TSA方法评估CRISPR-Cas9 RNP复合物形成的最佳条件》,该方案主要用于生物药品药物研发中临床前研究检测,参考标准--,《使用无标记TSA方法评估CRISPR-Cas9 RNP复合物形成的最佳条件》用到的仪器有NanoTemper PR Panta 多功能蛋白稳定性分析仪、NanoTemper PR Panta蛋白稳定性分析仪、PR-C002 | PR NT.48 系列标准毛细管
相关方案
更多
该厂商其他方案
更多












