“量体裁衣“ZFN,从源头降低岩藻糖!
“量体裁衣“ZFN,从源头降低岩藻糖!
前言
在理解锌指核酸酶之前,我们首先来回顾一下其他的核酸酶是如何切割特异性的DNA序列,从中我们可能会得到一些启示,如何对核酸酶进行改造,让它们能特异性地切割我们想要的序列。
限制性内切酶在原核生物中普遍存在,其主要目的是识别外源DNA并将其摧毁,从而抵御病毒的入侵。根据限制酶的结构,辅因子的需求切位与作用方式,可将限制酶分为三种类型:
Ⅰ型限制酶为复合功能酶,具有限制-修饰两种功能,但在 DNA链上没有固定的切割位点,一般在离切割位点1kb到几kb的地方随机切割,不产生特异性片段。Ⅲ型酶与Ⅰ型酶基本相似,不同的是Ⅲ型酶有特异性的切割位点,但这两类酶对DNA酶切分析的意义不大,通常所说的限制性内切酶是指Ⅱ型酶,它能够识别与切割DNA链上的特定的核苷酸顺序,产生特异性的DNA片段。
基因编辑三件套
剪刀
FokI是一种存在于细菌Flavobacterium okeanokoites的II型限制酶,由位于N端的DNA结合结构域(N-terminal DNA-binding domain)以及位于C端的非专一性DNA切割结构域(C-terminal DNA cleavage domain)组成。FokI的分子量为65.4 kDA,含有587个氨基酸。
和典型的II型限制酶一样,FokI核酸酶只要被招募到DNA上可以发挥功能,还有一个重要的特点就是,FokI核酸酶只有在二聚的情况下才能发挥功能。如下图所示,蓝色和绿色表示的两个FokI分子形成的二聚体结构,DNA recognition domain识别和结合位点是5’-GGATG-3’DNA序列,然后cleavage domain在距离识别位点下游的9-13个核苷酸的位置,对任意序列的核苷酸进行切割。
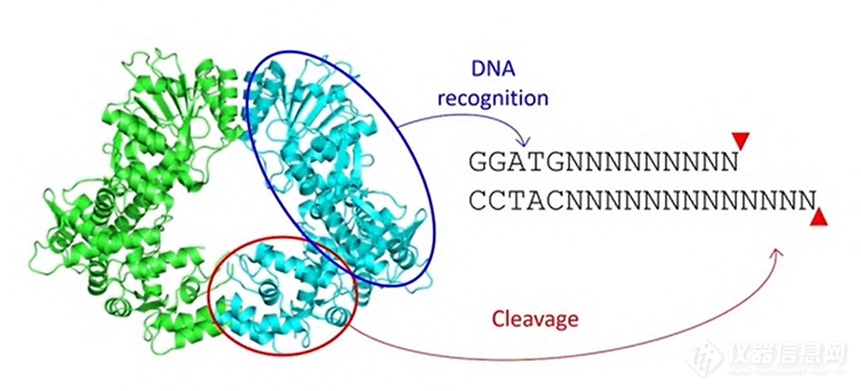
所以,我们在进行基因工程改造时,可以利用FokI核酸酶的cleavage domain,然后将其组装到其他的recognition domain上,就可以对特定的序列进行切割。
定位
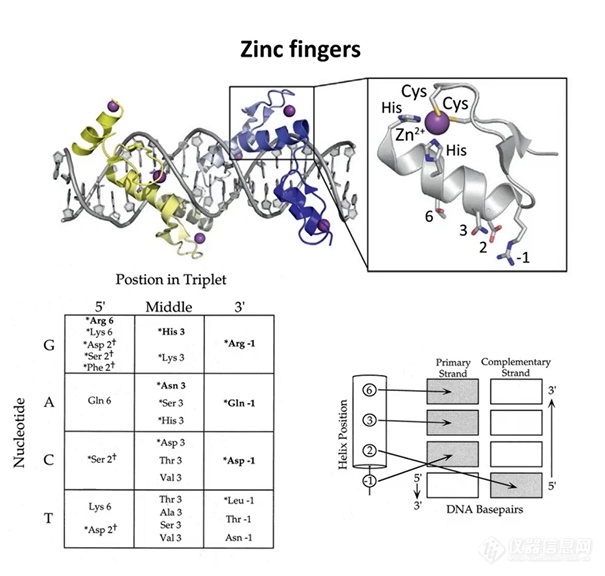
锌指首先在Aaron Klug实验室的爪蟾非洲爪蟾的转录研究中发现,是一种小的蛋白质motif,其特征在于结合一个或多个锌离子(Zn2+)以稳定折叠,由于结构类似手指而由此得名。在真核生物中从酵母到人类广泛存在,在人类的基因组中近1500种基因里包含锌指结构。根据残基的数量和顺序对不同类型的锌指进行分类(例如,Cys2His2, Cys4, 和 Cys6),其中最常见的是Cys2His2 ZFs。
锌指结构在1985年被发现,在1990s时期被深入研究,通过比较锌指蛋白的DNA识别区域的氨基酸序列以及特异性结合的DNA序列,得到以下结果:DNA识别域是由一系列 Cys2-His2锌指蛋白串联组成,其共有序列为(F/Y)-X-Cys-X2−5-C-X3-(F/Y)-X5-ψ-X2-His-X3−5-His,其中X为任意氨基酸,ψ是疏水性残基,形成一个简单的ββα折叠,这个锌离子位于反平行的β折叠和α螺旋之间,并且与β折叠中的两个半胱氨酸残基和α螺旋螺旋羧基端部分的两个组氨酸残基形成四配位化合物,如下图所示,这是锌指蛋白和DNA结合的三维结构图,锌指蛋白是结合在双链DNA的大沟处,每个锌指蛋白识别并结合3′到5′方向DNA链上一个特异的三联体碱基以及5′到3′方向的一个碱基。而锌指蛋白和DNA识别的特异性是取决于特定位置上的氨基酸。氨基酸的5′到3′方向上第6,3,2,-1号位置的不同的氨基酸分别对应结合的碱基,我们可以设计不同的氨基酸序列组合从而结合特定的DNA序列。例如5′-Arg6-His3-Ser2-Arg-1-3′这段氨基酸序列结合的DNA序列为5′-G-G-G-C-3′。
缝合
识别由锌指部分完成,切割由FokI核酸酶的cleavage domain完成,那切割后的DNA片段如何形成完整DNA序列呢?这就要引出细胞内部本身的一套系统--DNA修复机制,和用于定位的锌指蛋白、发挥剪刀功能的FokI核酸酶一样,DNA修复机制在细胞里已经存在了亿万年,自然也不是专门为我们给基因编辑的任务准备的。它的首要使命是保证基因组DNA的完整性,保证哪怕历经严酷的环境挑战,生命的遗传物质都不会被轻易破坏。
当锌指核酸酶在基因组中找到合适的位置并进行剪切之后,细胞自身就可以完成缝缝补补的针线活。其中存在两种不同的缝补办法。如果仅仅对基因组DNA一剪了之,没有现成模板可利用的细胞只能用一种简单粗暴的办法进行修复--非同源末端连接(Non-homologous end-joining, NHEJ),就是找到两个DNA断点,不管三七二十一直接粘上就行。这样的好处是简单快捷,坏处是难免会出现不该有的序列错误。为了避免这种错误,细胞会在附近找找有没有序列相似的DNA分子,以它为模板进行修复。这样万一真的出现了不该有的丢失或增加,也可以及时改正。如果在剪切的同时主动给细胞提供一段可供参考的模板序列,细胞就会启动另一种修复机制--DNA的同源重组机制(DNA homologous recombination),按照模板的序列信息老老实实地修复断裂的DNA。
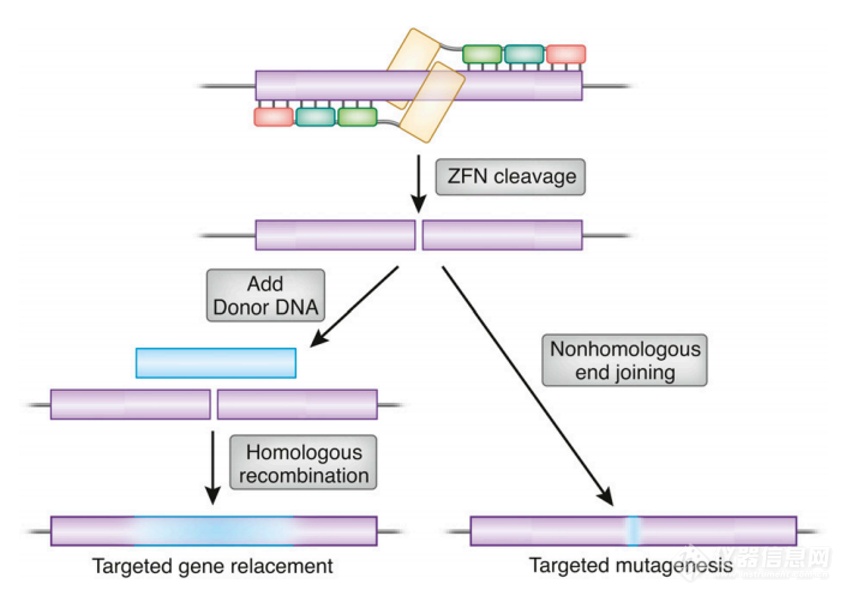
这两种缝补办法其实各有各的用处!如果我们想破坏掉一个正常基因的功能,就像破坏掉正常人体内的CCR5基因以期阻止HIV的入侵,前一种不够精确的缝补方法就可以帮助我们达到目的。如果我们想要修复一个原本就有问题的基因,就像镰刀形红细胞贫血症患者体内的HBS基因,那么后一种缝补方法正好能满足需求。
讲到这里,我们已经找到了精确基因编辑所需的工具三件套:
●基因组定位:锌指蛋白;
●基因组剪刀:FokI核酸酶的剪切模块;
●基因组缝合:细胞内天然存在的两套DNA修复机制;
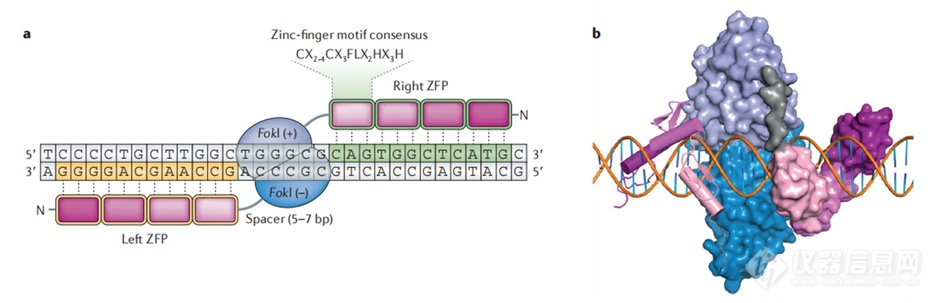
有了这三件套之后,我们就能在感兴趣的地方进行缝缝补补。针对靶序列设计若干个锌指结构域,将这些锌指结构域连在FokI核酸酶上,便可实现靶序列的双链断裂(Double strand break, DSB),最后利用DNA修复机制进行缝合。
基因编辑工具
ZFN剪辑有以下特点:
1.FokI cleavage domain 没有序列特异性,而且只能以二聚体的形式发挥功能。
2.对FokI cleavage domain 进行改造,减少脱靶效应。
3.需要构建两组ZFN结构,每组最佳为4个锌指结构,一共识别24个碱基对,增加了对靶序列识别的特异性。
除了锌指核酸酶,还有两种基因编辑工具也备受瞩目,那就是转录激活因子样效应因子核酸酶 (transcription activator-like effector nucleases, TALEN)和CRISPR/Cas 系统(CRISPR associated)。下面就简单介绍一下这两种基因编辑工具:
TALEN:和锌指核酸酶技术类似,科学家们发现了另一种DNA识别模块TALE蛋白, TALE蛋白是一种源自植物致病菌——黄单包杆菌(Xanthomonas)的天然蛋白,其中也含有DNA结合结构域。由于TALE具有序列特异性结合能力,研究者通过将FokI核酸酶与一段人造TALE连接起来,形成了一类具有特异性基因组编辑功能的强大工具,即TALEN。自2010年正式发明 TALEN技术以来,全球范围内多个研究小组利用体外培养细胞、酵母、拟南芥、水稻、果蝇及斑马鱼等多个动植物体系验证了TALEN的特异性切割活性。近年来,随着TALEN技术逐渐成熟,全球范围内各实验室已广泛使用TALEN技术来 完成基因打靶操作,但是该技术主要用于科研用途,暂时没有商业化产品可以使用。
CRISPR/Cas 系统:CRISPR/Cas作为原核生物中普遍存在的一种免疫系统,最初的功能就是识别外源性入侵的核酸序列,并对其进行特异性降解,以达到抗病毒的作用。其中CRISPR由一系列高度保守的重复序列(repeat)与同样高度保守的间隔序列(spacer)相间排列组成。CRISPR区域第一个重复序列上游有一段CRISPR的前导序列(Leader sequence),该序列作为启动子来启动后续CRISPR序列的转录,转录生成的RNA被命名为CRISPRRNA(简称crRNA)。CRISPR/Cas系统中crRNA与tracrRNA(反式激活的crRNA)形成嵌合RNA分子,即单向导RNA(Single guide RNA,sgRNA)。sgRNA可以介导Cas9蛋白在特定序列处进行切割,形成DNA双链断裂(Double-Stranded Break, DSB),完成基因定向编辑等的各类操作。CRISPR/Cas 系统现有基因编辑和基因修饰里面效率最高、最简便、成本最低、最容易上手的技术之一,成为当今最主流的基因编辑系统。2017年2月15日,美国专利局正式宣布,认可了哈佛大学-麻省理工学院博德研究所(Broad Institute)张锋团队拥有其开发的CRISPR/Cas9基因编辑工具的专利权,所以现在如果商业化使用该技术,需要支付高昂的专利费。
ZFN——商业化首选
虽说对ZFN的研究已经很深入,但是对于大部分研究人员来说,如果想要设计两组ZFN蛋白对靶序列进行切割,需要大量的实验探索,因为如前文所描述,每一根锌指并不是完美对应3碱基序列,手指会稍微“长”那么一点点,覆盖到前后的DNA碱基上。因此,当把几根锌指串联起来时,就有可能存在互相干扰,如果想设计出靶向目标序列且不互相干扰的锌指结构,是天文数字般的手指组合以及漫长的信息积累过程。哪些手指组合值得留意?到底怎么计算两根手指的相似性?这些问题筑起了高高的技术壁垒,直到今天,筛选组装出一套好用的锌手指也不是一件轻而易举的事情。
ZFN技术由Sangamo生物科学公司所拥有,被用来开发基因及细胞治疗产品。走在最前面的是和辉瑞合作开发的治疗A型血友病的基因疗法,已经进展到三期临床,还有3个基因和细胞治疗产品进展到1/2临床,十多个产品在临床前阶段,涵盖了罕见病、神经性疾病、肿瘤疾病及自身免疫疾病等多个方面,目标是开发出改变患者生活的基因组药物。

不过,对于ZFN在基因编辑的商业应用,Sangamo则授权给了Sigma-Aldrich,随着sigma被Merck收购,ZFN专利所有权属于Merck。公司投入了大量资金用于研究ZFN算法,最终推出了CompoZr 锌指核酸酶产品,Merck提供定制锌指核酸酶服务,您可扫描文末二维码留言,我们将尽快和您取得联系。
除了定制化的锌指核酸酶服务,Merck也提供一些目录产品。谷氨酰胺合成酶(GS)是生物制药行业最常用的可选标记之一。谷氨酰胺是细胞生长所必需的氨基酸。GS酶负责谷氨酸转化为谷氨酰胺。没有这种酶活性,细胞就不能再内源性合成谷氨酰胺,如果培养基中没有补充谷氨酰胺,细胞就会死亡。CHOZN® GS-/-细胞株利用Merck公司的专利技术CompoZr锌指核酸酶(ZFN)技术在亲本CHO-K1细胞中进行基因修改造,敲除了谷氨酰胺合成酶(GS),形成GS缺陷型的CHOZN® GS-/-细胞系,目前这个细胞系已经在国内外生物医药市场广泛应用。
更多![]()
使用高剪切湿法制粒工艺充分发挥Delta晶型甘露醇的优势
厂商
2022.04.06
一个“金牌”元件出马,载体“更高更快更强大”
厂商
2022.04.06
宿主细胞使用的技术梳理
厂商
2022.04.05
默克完成收购生物制药CDMO公司Exelead,并计划投资逾5亿欧元提升技术能力
新品
2022.04.05












